アンチセンス核酸の設計
アンチセンス核酸を含む核酸医薬は、疾患に関連する遺伝子の配列情報を基に、誰でも設計することが可能という特徴があります。
しかしながら、実際に効果の高い配列を設計するにはいくつかのポイントがあることが分かっています。
本稿では、アンチセンス核酸の設計方法について、これまで知られている設計のポイントを説明いたします。
1. アンチセンス核酸の分子構成と合成
当ホームページ「アンチセンス核酸の合成」でも示しました通り、投与後の分解を防ぎ、アンチセンス核酸としてオリゴヌクレオチドを機能させるためにはヌクレアーゼ耐性が必須です。
このため、DNAオリゴヌクレオチドを全てホスホロチオエート(PS)化し、PSオリゴとして合成します。
さらにPSオリゴに対し、ターゲットのRNAに対する親和性を高める目的で、核酸アナログである2’OMe, 2’MOE, LNAを導入する場合もあります。
この時、核酸アナログはアンチセンス分解機構を構成するRNase Hによって認識されないため、中央にRNase H認識ドメインとしてDNAを6~8塩基配置し、両端に核酸アナログを配置します。このようなアンチセンス核酸をGapmerアンチセンスと呼びます。
両端の核酸アナログ部が分解を緩和、RNAへの親和性を強化するため、高いアンチセンス効果が得られると言われています。
また、RNase H活性に依存しないアンチセンス核酸の場合は位置を問わずに核酸アナログを混ぜて合成すること(Mixmerアンチセンス)も可能です。
この場合はDNAが連続しないよう核酸アナログを混ぜて合成するか、全て核酸アナログとして合成を行います。
核酸医薬に使用される核酸分子・修飾
| 核酸アナログ・修飾核酸 | 修飾部位 | RNase H認識 | ターゲットへの親和性 | 備考 |
|---|---|---|---|---|
| ホスホロチオエート化 | リンケージ | あり | 低い | 高いヌクレアーゼ耐性 |
| 2’OMe | 糖 | なし | 高い | 天然型修飾RNA |
| 2’MOE | 糖 | なし | 高い | 毒性低 |
| LNA | 糖 | なし | 高い | 親和性大幅向上、毒性高 |
| 5-MeC | 塩基 | あり | 高い | TLR9反応回避 |

もっと詳しく:アンチセンス核酸の合成
2. アンチセンス核酸の配列選択方法
① mRNAの二次構造予測を行う
アンチセンス核酸はターゲットとなるmRNAに相補的に結合することで作用することより、mRNAとの結合部位がステムなどのRNA二次構造を取っているかいないかで効率が大きく変化すると考えられます。
従って、ターゲット配列の二次構造を予測し、生理条件下で2本鎖ステム構造を取るような部分を避け、1本鎖ループ部分の配列に相補するよう設計します。
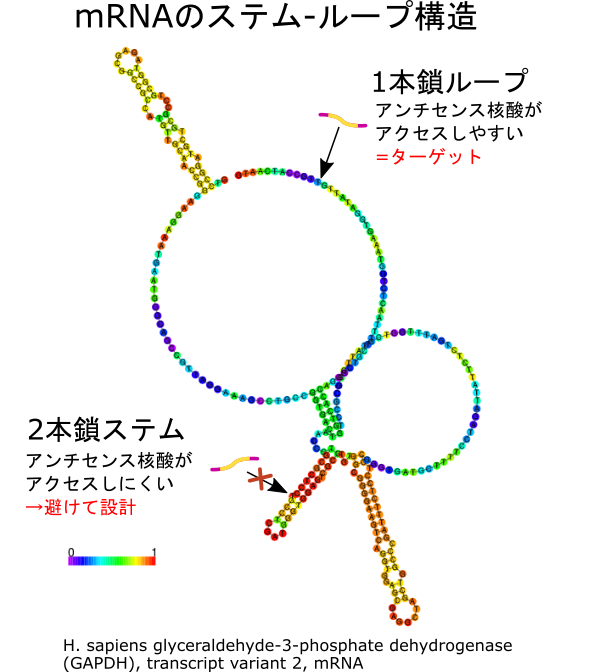
RNAの二次構造予測にはmfold(http://www.unafold.org/、結果は計算後6時間の間サイト上に公開)、RNAfold(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi)などのオンラインサイトを利用することが可能です。
また、RNase Hを用いてmRNAへのアクセシビリティーをマッピングし、実験的に結合効率の高い部位を推測する手法も取られます。
② 得られた標的配列のオフターゲットを確認する
標的とする配列が決定したら、標的外の遺伝子の中に、標的配列とのホモロジーが高くオフターゲットを示しうる遺伝子がないか確認します。
また、後のin vivo試験を見据え、ヒト以外に実験動物であるマウスなど他の哺乳動物についてもオフターゲットがないか確認することも重要です。
標的外の遺伝子と相同性を持ち、ハイブリダイゼーションを起こす狭義のオフターゲット効果は近年の研究で、特にLNAを含むGapmerアンチセンスの毒性に関連することが示されており、そのような配列は注意深く除外することが必要です。
③ オリゴがダイマー・高次構造を生じないかを確認する
有効かつ特異性の高い配列が得られたら、それを基にオリゴヌクレオチドの配列を設計しますが、その際オリゴヌクレオチドがダイマーを形成したり、オリゴ自身で高次構造を取らないかを確認します。
④ アンチセンス核酸のデザイン上避けるべき配列は
上①~③で示したポイントの他に、アンチセンス核酸のデザイン上避けるべき配列があります。
細菌由来のゲノムに特徴的な非メチル化CpGモチーフは脊椎動物の免疫刺激を引き起こすことが知られています。特にPu-Pu-CpG-Py-Py(Pu=プリン塩基、Py=ピリミジン塩基)モチーフは強力な免疫刺激を有するため、可能であればこのモチーフを回避して設計します。
このモチーフを避けることが難しい場合、シトシン塩基の代替として修飾塩基の5-メチルシトシンを用いて合成することによって、免疫応答を緩和させることが示されています。
5-メチルシトシンを含むオリゴ合成など、核酸医薬研究・配列スクリーニング・条件検討などにご利用いただける核酸合成サービスをご案内します
ニッポンジーン社 修飾DNAオリゴ合成サービス(グループ会社のホームページに移動します)
ニッポンジーンマテリアル 核酸医薬品研究・開発用オリゴヌクレオチド合成サービス
また、安定なDNA/RNA高次構造のG-quadruplexを形成するグアニン塩基リッチな配列(特にTTAGGG、TTGGGG)についても、避けたほうが良いと考えられます。
⑤ アンチセンス核酸の鎖長について
アンチセンス核酸の鎖長は一般的に12merから28mer、典型的には15merから22merの間で設定します。
25merを超えると細胞内への移行性が低下し、短い方がコスト上有利かつ移行性も良好ですが、短いほどハイブリダイゼーション可能なオフターゲットmRNA(狭義のオフターゲット)の数は増加し、副作用などの発現の可能性が上昇します。
オフターゲット効果は核酸医薬の毒性の度合いと相関すると言われているため、鎖長の選択はオフターゲット効果を考慮し、配列の最適化や核酸アナログの選択で回避できるか検討するなど戦略的に決定する必要があると考えられます。
FAQ
アンチセンス核酸のターゲット配列として、開始コドン領域は使用できますか?
ターゲットmRNAの2次構造を解析し、ループ部分であることを確認できれば、開始コドン部位はターゲット配列に使用できます。配列がループ部分で、かつリボソームや翻訳開始因子、スプライソソームなどmRNAの成熟・翻訳開始に関わるタンパク質複合体の結合部位として作用する部位であれば、アンチセンス核酸のターゲットとして使用することが可能と考えられます。
2’MOEとLNAをギャップマーに入れる際の注意事項を教えてください。
2’MOEとLNAはホスホロチオエート化(PS化)したギャップマーオリゴとしてアンチセンス核酸に多く用いられます。2’MOEはヌクレアーゼ耐性とターゲットへの親和性を向上させ、毒性を低減させる効果があります。LNAはよりヌクレアーゼ耐性と親和性が高い核酸アナログですが、毒性が生じる場合があります。ギャップマーとして合成する際の典型的なデザインとしては、2’MOEは5塩基の2’MOE+10塩基DNA+5塩基2’MOEで構成されます。LNAは3塩基のLNA+10塩基DNA+3塩基LNAで構成されることが多いようです。
